 | | PATHWAY: suba01100 | |
| Entry |
|
|---|
| Name |
Metabolic pathways - Subtercola endophyticus |
|---|
| Class |
|
|---|
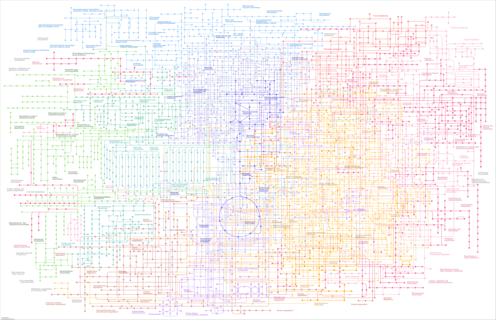
| Pathway map |
|
|---|
| Module |
| suba_M00007 | Pentose phosphate pathway, non-oxidative phase, fructose 6P => ribose 5P [PATH:suba01100] |
| suba_M00019 | Valine/isoleucine biosynthesis, pyruvate => valine / 2-oxobutanoate => isoleucine [PATH:suba01100] |
| suba_M00053 | Deoxyribonucleotide biosynthesis, ADP/GDP/CDP/UDP => dATP/dGTP/dCTP/dUTP [PATH:suba01100] |
| suba_M00125 | Riboflavin biosynthesis, plants and bacteria, GTP => riboflavin/FMN/FAD [PATH:suba01100] |
| suba_M00579 | Phosphate acetyltransferase-acetate kinase pathway, acetyl-CoA => acetate [PATH:suba01100] |
| suba_M00881 | Lipoic acid biosynthesis, plants and bacteria, octanoyl-ACP => dihydrolipoyl-E2/H [PATH:suba01100] |
| suba_M00909 | UDP-N-acetyl-D-glucosamine biosynthesis, prokaryotes, glucose => UDP-GlcNAc [PATH:suba01100] |
| suba_M00916 | Pyridoxal-P biosynthesis, R5P + glyceraldehyde-3P + glutamine => pyridoxal-P [PATH:suba01100] |
|
|---|
| Organism |
Subtercola endophyticus [GN: suba]
|
|---|
Related
pathway |
| suba00040 | Pentose and glucuronate interconversions |
| suba00130 | Ubiquinone and other terpenoid-quinone biosynthesis |
| suba00250 | Alanine, aspartate and glutamate metabolism |
| suba00260 | Glycine, serine and threonine metabolism |
| suba00280 | Valine, leucine and isoleucine degradation |
| suba00290 | Valine, leucine and isoleucine biosynthesis |
| suba00311 | Penicillin and cephalosporin biosynthesis |
| suba00361 | Chlorocyclohexane and chlorobenzene degradation |
| suba00400 | Phenylalanine, tyrosine and tryptophan biosynthesis |
| suba00514 | Other types of O-glycan biosynthesis |
| suba00520 | Amino sugar and nucleotide sugar metabolism |
| suba00525 | Acarbose and validamycin biosynthesis |
| suba00541 | O-Antigen nucleotide sugar biosynthesis |
| suba00625 | Chloroalkane and chloroalkene degradation |
| suba00630 | Glyoxylate and dicarboxylate metabolism |
| suba00660 | C5-Branched dibasic acid metabolism |
| suba00760 | Nicotinate and nicotinamide metabolism |
| suba00907 | Pinene, camphor and geraniol degradation |
| suba00999 | Biosynthesis of various plant secondary metabolites |
| suba01040 | Biosynthesis of unsaturated fatty acids |
| suba01059 | Biosynthesis of enediyne antibiotics |
|
|---|
| KO pathway |
|
|---|
| LinkDB |
|
|---|
|
|